Se descubre una actividad celular que sugiere que la función de reciclaje reside en nuestro ADN
Publicado originalmente por Rose Miyatsu, University of California - Santa Cruz, el 11 de mayo de 2024
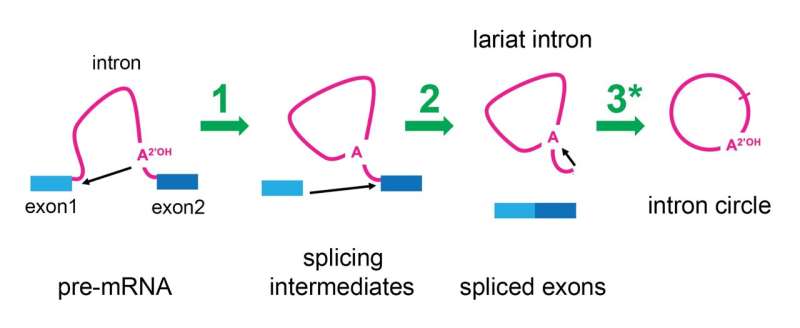
En la imagen se muestra la secuencia del splicing (cote y empalme). El ARN pre-mensajero (ARNpre-m) tiene exones (azul) e intrones (rosa). Se sabía que el espliceosoma (no se muestra) cataliza dos reacciones químicas (flechas negras) en un proceso de dos pasos (flechas verdes marcadas como 1 y 2) que empalma los exones y elimina el intrón como un lariat (estructura en lazo). Este estudio demuestra que, una vez finalizado el empalme, el espliceosoma sigue activo y puede convertir el intrón lariado en un círculo, mediante una tercera reacción (flecha verde 3) marcada con un asterisco. Crédito: Manuel Ares, UC Santa Cruz
Aunque tal vez no sea consciente de su existencia, o ni siquiera haya oído hablar de ellos, por todo su cuerpo trabajan duramente innumerables máquinas microscópicas llamadas espliceosomas. Mientras usted se sienta a leer, con gran fidelidad y rapidez están recomponiendo la información rota de sus genes, eliminando secuencias llamadas "intrones" para que sus ARN mensajeros puedan fabricar las proteínas correctas que necesitan sus células.
Los intrones son quizá uno de los mayores misterios de nuestro genoma. Son secuencias de ADN que interrumpen la información sensible que codifica proteínas en los genes, y necesitan ser "empalmados". El genoma humano tiene cientos de miles de intrones, unos 7 u 8 por gen, y cada uno de ellos es eliminado por un complejo especializado de proteínas de ARN llamado "espliceosoma" que corta todos los intrones y empalma las secuencias codificadoras restantes, llamadas exones. Se desconoce cómo evolucionó este sistema de genes rotos y el espliceosoma en nuestros genomas.
A lo largo de su dilatada carrera, Manny Ares, catedrático de Biología Molecular, Celular y del Desarrollo de la Universidad de Santa Cruz, se ha propuesto aprender todo lo que pueda sobre el empalme del ARN.
"Soy un apasionado del espliceosoma", afirma Ares. "Quiero saber todo lo que hace el espliceosoma, aunque no sepa por qué lo hace".
En un nuevo artículo publicado en la revista Genes and Development, Ares informa de un sorprendente descubrimiento sobre el espliceosoma que podría decirnos más sobre la evolución de las distintas especies y el modo en que las células se han adaptado al extraño problema de los intrones. Los autores demuestran que una vez que el espliceosoma ha terminado de empalmar el ARNm, permanece activo y puede tomar parte en otras reacciones en las que intervienen los intrones eliminados.
Este descubrimiento proporciona el indicio más sólido que tenemos hasta ahora de que los espliceosomas podrían ser capaces de reinsertar un intrón de nuevo en el genoma en otra localización. Se trata de una capacidad que anteriormente no se creía que poseyeran los espliceosomas, pero que es una característica común de los "intrones del Grupo II", primos lejanos del espliceosoma que existen principalmente en bacterias.
Se cree que el espliceosoma y los intrones del grupo II comparten un ancestro común responsable de la propagación de los intrones por todo el genoma, pero mientras que los intrones del grupo II pueden empalmarse a partir del ARN y volver directamente al ADN, los "intrones espliceosómicos" que se encuentran en la mayoría de los organismos superiores requieren el espliceosoma para empalmarse y no se creía que pudieran reinsertarse en el ADN. Sin embargo, el hallazgo del laboratorio de Ares indica que el espliceosoma podría seguir reinsertando intrones en el genoma en la actualidad. Se trata de una posibilidad interesante, ya que los intrones que se reintroducen en el ADN añaden complejidad al genoma, y comprender mejor de dónde proceden estos intrones podría ayudarnos a entender mejor cómo siguen evolucionando los organismos.
Un descubrimiento interesante
Los genes de un organismo están formados por ADN, en el que cuatro bases, adenina (A), citosina (C), guanina (G) y timina (T), se ordenan en secuencias que codifican instrucciones biológicas, como la fabricación de proteínas específicas que el cuerpo necesita. Antes de que se puedan leer estas instrucciones, el ADN se copia en ARN mediante un proceso conocido como transcripción, y luego hay que eliminar los intrones de ese ARN antes de que un ribosoma pueda traducirlo en proteínas reales.
El espliceosoma elimina los intrones mediante un proceso de dos pasos que hace que el ARN del intrón tenga uno de sus extremos unido al medio, formando un círculo con una cola parecida al "lazo" de un vaquero. Este aspecto ha llevado a denominarlos "intrones lazo". Recientemente, unos investigadores de la Universidad de Brown que estudiaban la ubicación de los puntos de unión de estos lazos hicieron una extraña observación: algunos intrones eran circulares en lugar de tener forma de lazo.
Esta observación llamó inmediatamente la atención de Ares. Algo parecía estar interactuando con los intrones lazo, después de ser eliminados de la secuencia de ARN para cambiar su forma, y el espliceosoma fue su principal sospechoso.
"Me pareció interesante por esta idea antigua sobre el origen de los intrones", dijo Ares. "Hay muchas pruebas de que las partes de ARN del espliceosoma, los snARN, están estrechamente relacionadas con los intrones del Grupo II".
Dado que el mecanismo químico de empalme es muy similar entre los espliceosomas y sus primos lejanos, los intrones del Grupo II, muchos investigadores han teorizado que cuando el proceso de autoempalme se volvió demasiado ineficiente para que los intrones del Grupo II pudieran completarse de forma fiable por sí solos, partes de estos intrones evolucionaron para convertirse en el espliceosoma. Mientras que los intrones del grupo II eran capaces de insertarse directamente en el ADN, se pensaba que los intrones espliceosómicos, que requerían la ayuda de los espliceosomas, no podían volver a insertarse en el ADN.
"Una de las preguntas que me faltaban en esta historia era: ¿es posible que el espliceosoma moderno siga siendo capaz de tomar un intrón lariat e insertarlo en algún lugar del genoma? dijo Ares. "¿Sigue siendo capaz de hacer lo que hizo el complejo ancestral?".
Para empezar a responder a esta pregunta, Ares decidió investigar si era realmente el espliceosoma el que realizaba cambios en los intrones lariat para eliminar sus colas. Su laboratorio ralentizó el proceso de empalme en células de levadura y descubrió que después de que el espliceosoma liberara el ARNm, del que había terminado de empalmar los intrones, se aferraba a los intrones lariados y les daba nueva forma, convirtiéndolos en verdaderos círculos. El laboratorio de Ares pudo volver a analizar los datos de secuenciación de ARN de células humanas publicados y descubrió que los espliceosomas humanos también tenían esta capacidad.
"Estamos entusiasmados porque, aunque no sabemos qué puede hacer este ARN circular, el hecho de que el espliceosoma siga activo sugiere que puede ser capaz de catalizar la inserción del intrón lariat de nuevo en el genoma", afirma Ares.
Si el espliceosoma es capaz de reinsertar el intrón en el ADN, esto también añadiría un peso significativo a la teoría de que los espliceosomas y los intrones del Grupo II compartieron un ancestro común hace mucho tiempo".
Comprobación de una teoría
Ahora que Ares y su laboratorio han demostrado que el espliceosoma tiene la capacidad catalítica de reinsertar hipotéticamente intrones en el ADN como hicieron sus antepasados, el siguiente paso consiste en que los investigadores creen una situación artificial en la que "alimenten" con una cadena de ADN a un espliceosoma que aún esté unido a un intrón lariat y comprueben si realmente consiguen que inserte el intrón en algún lugar, lo que supondría una "prueba de concepto" de esta teoría.
Si el spliceosoma es capaz de reinsertar intrones en el genoma, es probable que sea un evento muy infrecuente en los seres humanos, porque los spliceosomas humanos tienen una gran demanda y por lo tanto no pueden emplear mucho tiempo con intrones eliminados. Sin embargo, en otros organismos en los que el espliceosoma no está tan ocupado, la reinserción de intrones puede ser más frecuente. Ares colabora estrechamente con el catedrático de Ingeniería Biomolecular de la UCSC Russ Corbett-Detig, que ha dirigido recientemente una búsqueda sistemática y exhaustiva de nuevos intrones en los genomas disponibles de todas las especies que contienen intrones, que se publicó en la revista Proceedings of the National Academy of Sciences (PNAS) el año pasado.
El artículo publicado en PNAS mostraba que las "explosiones" de intrones en épocas remotas de la historia evolutiva probablemente introdujeron miles de intrones en un genoma de una sola vez. Ares y Corbett-Detig trabajan ahora para recrear artificialmente una explosión, lo que les permitiría saber cómo reaccionaron los genomas cuando se produjo.
Ares afirma que su colaboración interdisciplinar con Corbett-Detig les ha abierto las puertas a profundizar realmente en algunos de los mayores misterios de los intrones, que probablemente les sería imposible comprender plenamente sin su experiencia combinada.
"Es la mejor manera de hacer las cosas", afirma Ares. "Cuando encuentras a alguien que tiene en mente el mismo tipo de preguntas, pero un conjunto diferente de métodos, perspectivas, sesgos e ideas raras, la investigación se hace más emocionante y te lleva a creer que puedes abrirte camino y resolver un problema como éste, que es muy complejo".
Más información: Manuel Ares et al, Intron lariat spliceosomes convert lariats to true circles: implications for intron transposition, Genes & Development (2024). DOI: 10.1101/gad.351764.124
Información de la revista: Proceedings of the National Academy of Sciences, Genes & Development
Proporcionado por University of California – Santa Cruz



Comentarios
Publicar un comentario